Negli ultimi anni la ricerca sul miglioramento qualitativo dell’endomicroscopia tramite Intelligenza Artificiale ha visto il nostro gruppo contribuire sia con nuove metodologie che con studi sistematici. Di seguito tutti i lavori pubblicati principali, con una nota chiara su cosa apporta ciascun contributo.
Motivazioni & Obiettivi
Metodi e Risultati
Motivazioni & Obiettivi
- Elevare la qualità diagnostica delle immagini di endomicroscopia con tecniche di super-risoluzione, rendendo possibili applicazioni cliniche affidabili anche in assenza di dati ad alta risoluzione.
- Mettere a confronto, standardizzare e portare sul campo tecniche avanzate di deep learning per la SR e la ricostruzione spaziotemporale.
Metodi e Risultati
-
Adversarial training with cycle consistency for unsupervised super-resolution in endomicroscopy
Medical Image Analysis, 2019
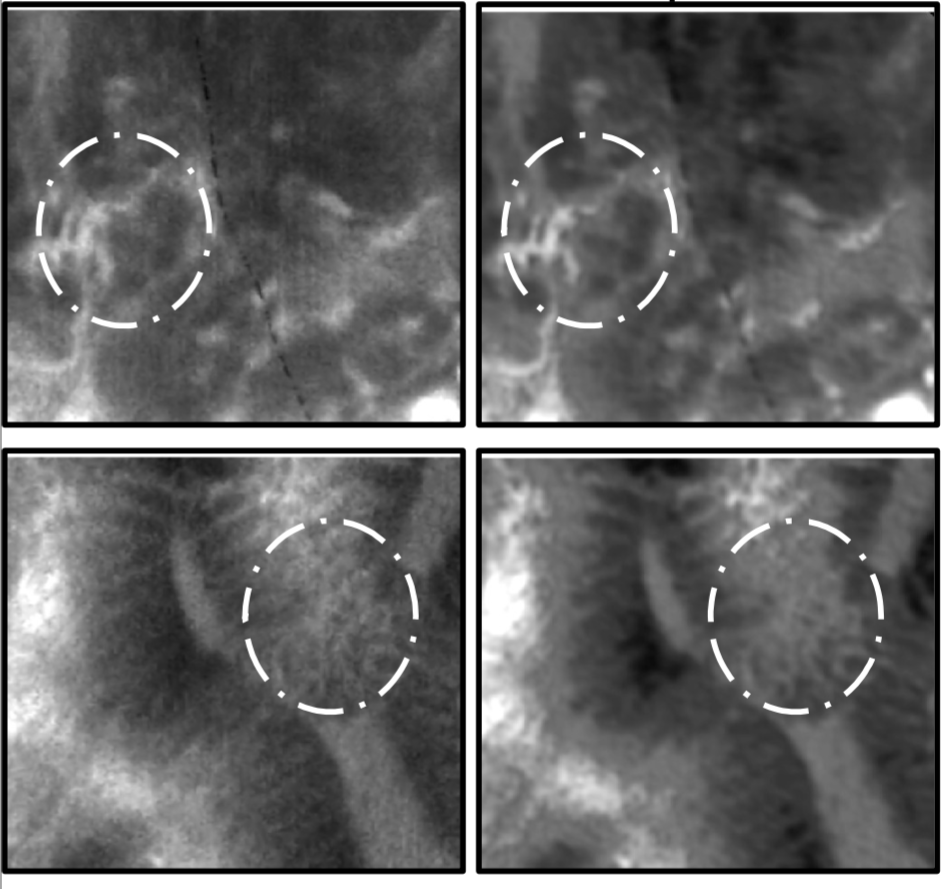
Primo framework non supervisionato basato su GAN e consistenza ciclica fisicamente ispirata: consente l’apprendimento della SR anche in assenza di coppie perfettamente allineate LR/HR, trasferendo la qualità HR tra domini diversi. Validato su 238 sequenze video da 143 pazienti, con metriche quantitative e studio MOS clinico. -
Effective deep learning training for single-image super-resolution in endomicroscopy exploiting video-registration-based reconstruction
International Journal of Computer Assisted Radiology and Surgery, 2018 – PMC Open Access
Propone una strategia efficace di generazione dati sintetici tramite video-mosaicking per addestrare DNNs allo SR, anche dove le immagini HR reali mancano. Analizza tre tecniche state-of-the-art su oltre 8800 immagini e conferma (anche con MOS) il guadagno qualitativo. -
Learning from irregularly sampled data for endomicroscopy super-resolution: a comparative study of sparse and dense approaches
International Journal of Computer Assisted Radiology and Surgery, 2020
Studio comparativo su CNN dense vs sparse per la super-risoluzione da input irregolari (tipici di pCLE); introduce anche strati kernel Nadaraya-Watson nel framework profondo e genera dati sintetici per benchmarking robusto.
Codice Repository
-
Al momento, il codice open-source specifico per la super-resolution endomicroscopica non è disponibile in repository pubblici dedicati.
Per richieste o collaborazioni, contattare gli autori dei lavori citati.
Team & Autori
- Daniele Ravì (PI, metodologie deep learning, progettazione framework GAN/SR, validazione su dati reali)
- Agnieszka B. Szczotka (idea e sviluppo sparse/dense CNN, dataset, validazione UCL)
- Stephen P. Pereira, Tom Vercauteren, D.I. Shakir, M.J. Clarkson (KCL, UCL, collaborazione clinica e supervisione)