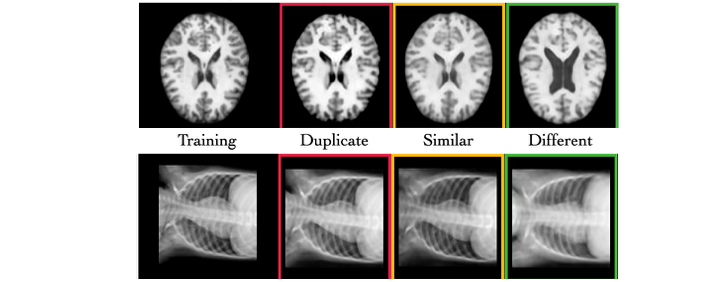
La diffusione di modelli generativi profondi ha portato alla creazione di dati sintetici per l’imaging medico, essenziali per ricerca e sviluppo di nuove tecniche diagnostiche. Tuttavia, questi modelli rischiano seriamente di “memorizzare” dati sensibili dei pazienti, generando immagini troppo simili ai dati di training ed esponendo quindi informazioni riservate. Il progetto DeepSSIM nasce per affrontare proprio questa criticità, proponendo una nuova metrica automatica e auto-supervisionata per quantificare quanto un modello generativo memorizzi i dati originali durante la sintesi.
Motivazioni & Obiettivi
Metodi
Impatto & Risultati
Motivazioni & Obiettivi
- Proteggere la privacy dei pazienti nell’era dell’AI medicale.
- Fornire uno strumento pratico per valutare il rischio di memorizzazione nei modelli generativi utilizzati in ambito sanitario.
- Promuovere una cultura della trasparenza e della riproducibilità nella ricerca su dati sensibili.
Metodi
- DeepSSIM proietta le immagini in uno spazio di embedding neurale apprendimento, ottimizzando la similarità coseno per riflettere il vero SSIM (Structural Similarity Index) tra immagini mediche.
- L’algoritmo sfrutta augmentations che preservano la struttura anatomica, consentendo di stimare la similarità senza la necessità di un perfetto allineamento spaziale.
- Validato su immagini sintetiche MRI generate da un Latent Diffusion Model allenato in condizioni ad alto rischio di memorizzazione.
Impatto & Risultati
- DeepSSIM supera tutte le metriche precedenti con un incremento medio F1 score del +52,03% nel rilevamento di memorizzazione.
- Garantisce analisi efficiente e scalabile anche su grandi dataset, velocizzando di decine di volte i calcoli rispetto all’SSIM tradizionale.
- Codice open-source disponibile per la comunità, favorendo trasparenza e applicazioni future.
Articoli Scientifici Correlati
-
A Novel Metric for Detecting Memorization in Generative Models for Brain MRI Synthesis
arXiv preprint arXiv:2509.16582 (2025) – Google Scholar
Codice Repository
- Repository ufficiale: brAIn-science/DeepSSIM
Team & Autori
- Antonio Scardace (Primo autore, sviluppo metodologico)
- Lemuel Puglisi (Co-autore, implementazione)
- Daniele Ravì (Supervisione scientifica)